Una faccia, una razza?
Fabio Fantini
Per stemperare il comprensibile clima di ostilità da parte delle popolazioni locali che accompagnava l’occupazione militare della Grecia, le autorità fasciste italiane coniarono il detto del titolo, privo del punto interrogativo, a volere bonariamente (nelle intenzioni) sottolineare che in fondo gli Italiani erano sì occupanti, ma non tanto stranieri. Se l’occupazione fu di breve durata, il detto ha avuto una vita assai più lunga e ancora oggi riecheggia nei porti mediterranei, professione pacificatoria di presunta comunanza delle origini, usato non solo tra Greci e Italiani.
Il detto suscita due interrogativi: se la somiglianza dei volti sia un indice attendibile delle relazioni di parentela etnica; se esistano limiti netti di diversità morfologica oltre i quali il detto perda di applicabilità, tanto da fare considerare due facce sufficientemente diverse da non potere più essere ascritte alla stessa razza. Come probabilmente è noto, la genetica, che non si perde dietro discussioni metafisiche, è in grado di dare risposte nette ai due interrogativi: no e no.
Le risposte della genetica sono quantitative e si basano su misurazioni più precise di quelle costruite sulla morfologia facciale, perché prendono in considerazione le sequenze nucleotidiche delle molecole ereditarie. Queste sequenze sono costituite da entità identificabili, numerabili e confrontabili, non sono influenzate da interazioni con l’ambiente che possano interferire con la manifestazione fenotipica del carattere e rappresentano i più attendibili testimoni delle nostre relazioni genealogiche.
Negli ultimi trenta anni il sequenziamento del DNA si è gradualmente trasformato da operazione lenta, ardua e costosa in un lavoro di routine. La crescente estensione delle banche dati disponibili per le caratteristiche genetiche delle popolazioni umane ha reso possibile effettuare analisi statistiche sempre più attendibili sulla distribuzione geografica della variabilità dei caratteri nella specie umana.
Nel 2002 un gruppo di ricercatori dell’Università di Stanford guidato da Noah Rosenberg pubblicò un’indagine riguardante 1056 persone provenienti da 52 aree geografiche diverse, nelle quali furono esaminate mutazioni a singolo nucleotide (SNP) in 377 loci situati in microsatelliti autosomici (STR). I dati furono elaborati grazie a un software che analizza le similarità per raggrupparle in un numero K di categorie, scelto dal programmatore. In altre parole, il software raggruppa le varianti SNP in un numero impostato di categorie, in modo che ciascuna categoria abbia la massima omogeneità al proprio interno e la massima disomogeneità con le altre categorie.
Il genoma di ogni individuo è considerato come il risultato dei contributi genetici provenienti da un numero K di popolazioni ancestrali, descritti da K coefficienti di ascendenza. Per ciascun individuo la somma dei coefficienti di ascendenza è 1. Nella rappresentazione grafica dei risultati, a ogni individuo corrisponde una linea verticale suddivisa in segmenti colorati, le cui lunghezze corrispondono al coefficiente di ascendenza relativo a ciascuna delle K categorie in cui si è deciso di raggruppare le ascendenze. Le linee verticali sono disposte una accanto all’altra tenendo conto dell’origine etnica e formano un dendrogramma, cioè una rappresentazione grafica dei coefficienti di similarità che si ottengono con un processo di analisi per raggruppamenti.
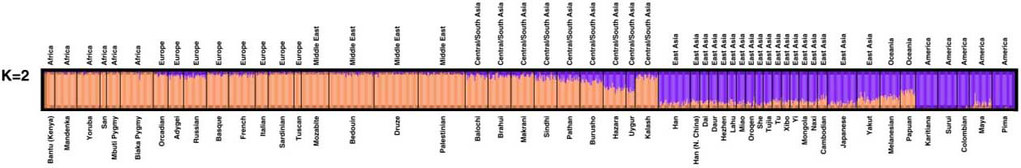
Un primo esempio di dendrogramma è fornito dalla figura 1. In questa figura compaiono due soli colori, cioè ogni linea che rappresenta un singolo individuo è costituita da segmenti solo di colore arancione o viola, perché il programma è stato impostato per raggruppare i dati sui genomi in due sole categorie (K= 2). Tutte le linee hanno la stessa altezza, corrispondente alla somma 1 dei coefficienti di ascendenza relativi ai due gruppi in cui il programma ha suddiviso gli SNP relativi ai 377 loci esaminati. Ogni linea è costituita da segmenti arancioni o viola o in parte arancioni e in parte viola.
La successiva figura 1 offre un colpo d’occhio immediato di come potremmo suddividere l’umanità, sevolessimo ascriverla al contributo genetico di due sole popolazioni ancestrali. Gli individui delle popolazioni africane, europee, mediorientali e dell’Asia centromeridionale hanno una larga dominanza del colore arancione, mentre in quelli delle popolazioni dell’Estremo Oriente, dell’Oceania e delle Americhe prevale il colore viola.
Il risultato di questa prima operazione di raggruppamento indica la presenza di un gruppo di popolazioni del Continente Antico (Africa, Eurasia) distinto dal secondo gruppo, presente nelle aree investite solo in un secondo tempo dall’ondata di espansione delle popolazioni umane moderne.
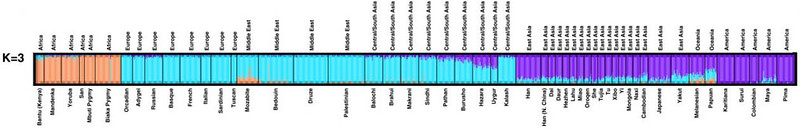
Il dendrogramma di figura 2 è ottenuto impostando a tre il numero dei raggruppamenti e quindi delle popolazioni ancestrali e vede pertanto l’ingresso di una terza categoria, indicata con il colore celeste. La distribuzione geografica dei tre raggruppamenti è grossolanamente riportabile a un nucleo africano, a un nucleo europeo e asiatico centromeridionale, a un terzo gruppo diffuso prevalentemente in Estremo Oriente, in Oceania e nelle Americhe.
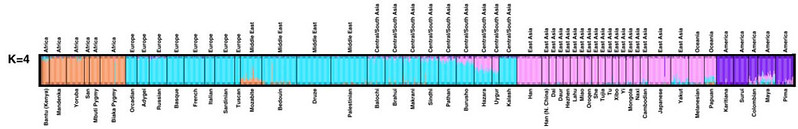
Se le categorie salgono a quattro (figura 3), il nuovo raggruppamento che emerge, identificato dal colore rosa, corrisponde alle popolazioni dell’Estremo Oriente e dell’Oceania, che si separano da quelle delle Americhe. Appartengono a questo raggruppamento anche componenti genetiche minoritarie presenti nelle popolazioni dell’Asia centromeridionale.
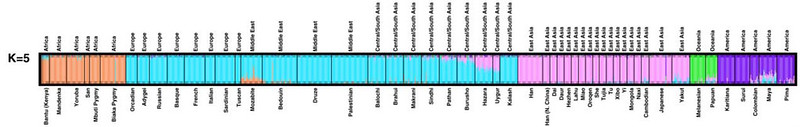
Il dendrogramma di figura 4 rappresenta la suddivisione in cinque raggruppamenti, che segna la separazione delle popolazioni dell’Oceania, indicate con il prevalente colore verde, e riproduce, per grandi linee, la distribuzione geografica continentale delle popolazioni umane, apparente conferma dell’esistenza dei gruppi razziali tradizionali su base geografica continentale, vale a dire Africani, Indoeuropei, Asiatici orientali, Melanesiani, Amerindi.
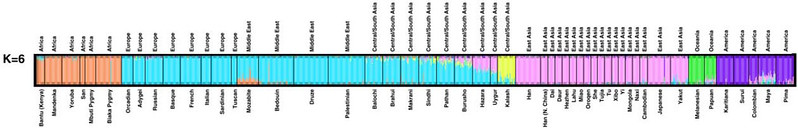
Se però le suddivisioni diventano sei (figura 5), il nuovo gruppo che si forma, indicato con il colore giallo, è quello dei Kalash, una tribù di circa 4000 persone che vive nel Pakistan settentrionale, un popolo con caratteristiche insolite, ma non così insolite da guadagnarsi un raggruppamento razziale distinto.
Ci si può chiedere che cosa succederebbe se portassimo a sette le aggregazioni, con il dubbio che potrebbe emergere qualche popolazione ancora più piccola dei Kalash. Ma non è così: il passaggio a sette raggruppamenti (figura 6, separata in due mezze strisce per facilitare la lettura) vede la divisione del grande gruppo indoeuropeo in due ammassi consistenti, europei e mediorientali da una parte e asiatici meridionali dall’altra (colore rosso).
Si può continuare ad aumentare il numero dei raggruppamenti e si osserva un aumento dei gradienti nelle zone di confine tra i gruppi. Il maggior numero di somiglianze si riscontra all’interno di un gruppo, ma molte caratteristiche si sovrappongono con altri gruppi, in particolare quelli geograficamente più prossimi. La variabilità umana diventa un continuum ininterrotto, nel quale porre confini divisori tra un gruppo e un altro è un’operazione puramente arbitraria.
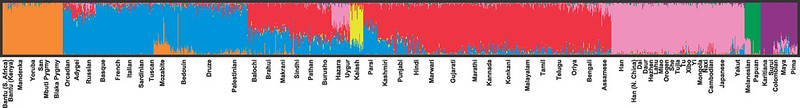
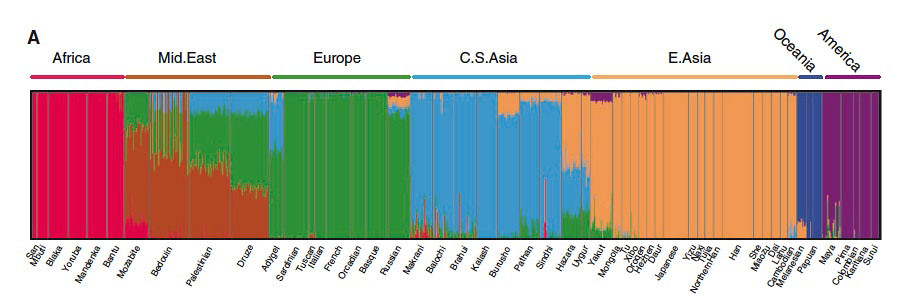
Nel 2008 un gruppo di ricercatori sempre dell’Università di Stanford, comprendente tra gli altri anche Luigi Luca Cavalli Sforza, ripeté uno studio simile a quello eseguito da Rosenberg e colleghi. Nel frattempo le tecniche di indagine avevano subito un notevole progresso, tanto che in questo secondo studio furono presi in considerazione 650.000 SNP in 938 individui appartenenti a 51 popolazioni. L’analisi per raggruppamenti ottenuta sulla base di questa maggiore quantità di dati è rappresentata nel dendrogramma di figura 7, nel quale il numero dei raggruppamenti è stato impostato a sette. I colori impiegati sono diversi da quelli dei dendrogrammi costruiti da Rosenberg e collaboratori, ma il quadro complessivo è sostanzialmente invariato.I dati raccolti nello studio del 2008 sono stati utilizzati anche per costruire un albero filogenetico delle popolazioni umane secondo il criterio della massima parsimonia.
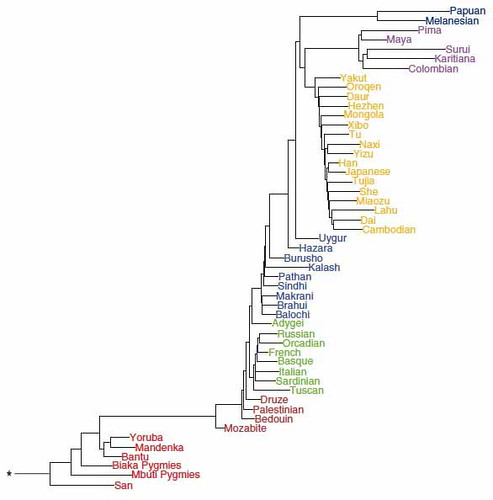
Lo schema di figura 8 presenta questo albero, nel quale i nomi delle popolazioni sono scritti con gli stessi colori della figura 7 per indicare la provenienza geografica.
La diversità genetica della specie umana è massima in Africa e decresce man mano che ci si allontana da questo continente, probabilmente a causa di ripetuti effetti del fondatore che hanno segnato l’espansione dei sapiens moderni negli altri continenti. Come risultato, gli alleli umani hanno per la maggior parte una distribuzione cosmopolita, presenti come sono in tutti i continenti, anche se con frequenze diverse. Lo studio degli aplotipi consente una caratterizzazione migliore, perché hanno una distribuzione geografica più chiara; e tuttavia solo una minoranza degli aplotipi risulta specifica di un solo continente.
Con una popolazione che supera abbondantemente i sette miliardi di individui sparsi su un areale ampio quanto la quasi totalità delle terre emerse, ci si potrebbe attendere un’ampia variabilità genetica nelle popolazioni umane. Il fatto che non sia così fa pensare che la maggior parte della storia delle popolazioni umane sia stata caratterizzata da popolazioni di dimensioni ridotte. Inoltre, gli umani sono attivamente mobili, distribuiti in modo sostanzialmente continuo sulle masse continentali (esclusa l’Antartide) e fecondi tutto l’anno.
Non c’è troppo da meravigliarsi se i flussi genici tra le diverse popolazioni sono stati consistenti e frequenti per tutta la storia dell’umanità. È sufficiente ricordare gli incroci con popolazioni neandertaliane e denisovane avvenuti nel tardo Paleolitico, per rammentare la facilità con la quale possono avvenire rimescolamenti del patrimonio ereditario grazie alla costante propensione, assai diffusa nella specie umana e probabilmente anche nelle estinte specie congeneri, all’accoppiamento sessuale. In rapporto agli altri primati, la specie umana è geneticamente molto omogenea: se scegliamo a caso due individui della nostra specie, in media a differire è solo un nucleotide ogni mille.
Non si può proibire per decreto di parlare di razze umane, basta si abbia chiaro che il termine non ha significato biologico. Se si vuole usare questo termine carico di un pesante fardello storico, al posto dei più appropriati etnia o popolazione, nulla vieta di farlo: l’ignoranza culturale non è perseguibile per legge.
Parlare di razze biologiche umane è un’imprecisione. Richiamarsi a concetti quali «purezza della razza» è invece una barzelletta o, se l’affermazione è fatta con serietà, un errore che rivela non solo la pervicace nostalgia di politiche criminali del passato, ma anche la negligente disattenzione per le conoscenze scientifiche. Se, negli anni Cinquanta del Novecento, l’ipotesi multiregionalista sostenuta con grande impegno dall’antropologo americano Carleton Coon poteva essere considerata come un modello opinabile ma comunque degno di essere messo alla prova, i dati che si sono accumulati negli ultimi sessanta anni non lasciano dubbi sull’origine africana, recente e unica dell’umanità attuale.
Se è vero che nella popolazione umana esistono alcune caratteristiche morfologiche e comportamentali distribuite in modo discontinuo, ciò non autorizza a sostenere una visione frammentata dell’umanità sulla base di inesistenti considerazioni biologiche, dalle quali fare discendere, in sovrappiù, considerazioni su tipologie di qualità comportamentali e intellettuali. Il concetto di razza è inapplicabile allo schema globale della diversità genetica umana. Le tassonomie razziali non sono nulla di più che comode categorizzazioni per incasellare senza fatica le persone in uno schema precostituito. Si tratta di una comprensibile ma pericolosa scorciatoia per semplificare la classificazione di una umanità varia e imprevedibile e per affermare una propria identità in un contesto sociale che ci rende sempre più insicuri, soprattutto quando siamo privi di una visione culturale articolata e profonda.
Bibliografia
Li J. Z., Absher D. M., Tang H., Southwick A. M., Casto A. M., Ramachandran S., Cann H. M., Barsh G. S., Feldman M., Cavalli-Sforza L. L., Myers R. M., Worldwide Human Relationships Inferred from Genome-Wide Patterns of Variation, Science 22 Feb 2008, Vol. 319, Issue 5866, pp. 1100-1104, scaricabile al sito https://www.researchgate.net/publication/5560546_Worldwide_Human_Relationships_Inferred_from_Genome-Wide_Patterns_of_Variation
Rosenberg N. A., Pritchard J. K., Weber J. L., Cann H. M., Kidd K. K., Zhivotovsky L. A., Feldman M., Genetic structure of human populations, Science 20 Dec 2002, Vol. 298, Issue 5866, pp. 2381-2385, scaricabile al sito https://rosenberglab.stanford.edu/papers/popstruct.pdf